Defensa insólita contra virus bacteriófagos: síntesis de un gen nuevo… y tóxico
Francis Crick, codescubridor de la doble hélice, definió como “dogma central de la Biología Molecular” el hecho de que la información genética fluya en una sola dirección, del ADN al ARN mensajero y de este último a la secuencia de aminoácidos en la proteína. Este dogma tuvo que ser revisado cuando se descubrieron las retrotranscriptasas […] El artículo Defensa insólita contra virus bacteriófagos: síntesis de un gen nuevo… y tóxico se ha escrito en Cuaderno de Cultura Científica.
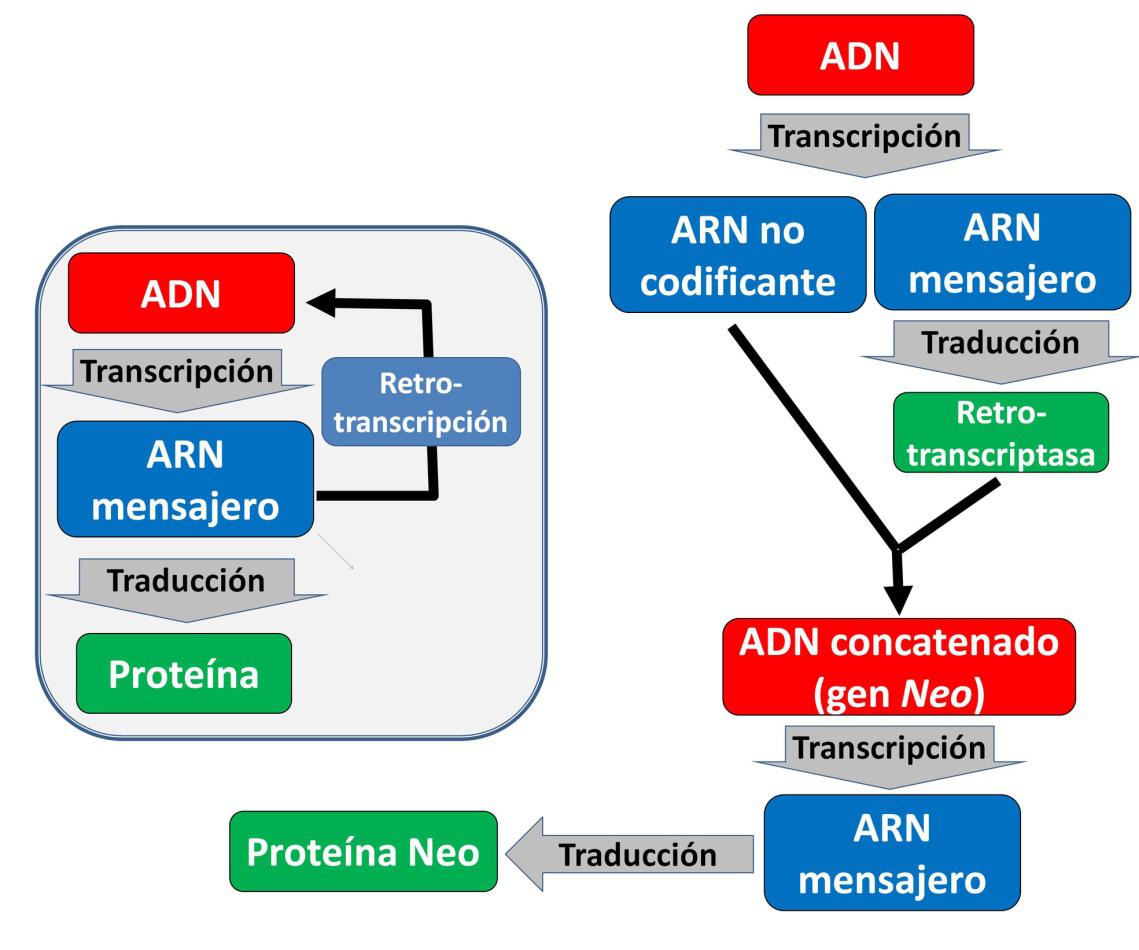
Francis Crick, codescubridor de la doble hélice, definió como “dogma central de la Biología Molecular” el hecho de que la información genética fluya en una sola dirección, del ADN al ARN mensajero y de este último a la secuencia de aminoácidos en la proteína. Este dogma tuvo que ser revisado cuando se descubrieron las retrotranscriptasas o transcriptasas inversas, enzimas capaces de sintetizar ADN a partir de secuencias de ARN (Figura 1). Estas enzimas son producidas por retrovirus como el VIH-1 para insertar su información en el ADN de la célula infectada. También nosotros utilizamos la retrotranscriptasa para amplificar las secuencias de ADN conocidas como retrotransposones y trasladarlas de una parte del genoma a otra. De hecho, más del 40% de nuestro genoma está constituido por retrotransposones.
Las bacterias también utilizan las retrotranscriptasas como medio para defenderse de los virus bacteriófagos, sus grandes enemigos naturales. Esta estrategia defensiva suele activarse cuando han fallado los mecanismos basados en la degradación del material genético vírico. Un ejemplo de estos mecanismos es el conocido sistema CRISPR-Cas, que en la actualidad constituye una tecnología sencilla y eficaz para la edición genética.
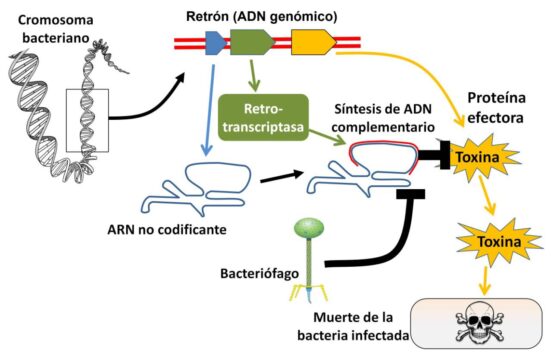
Si la infección vírica desborda esta primera línea de defensa, se activan procesos basados en retrotranscriptasas, descubiertos recientemente y mucho menos conocidos. Algunos de estos mecanismos defensivos se denominan retrones. Los retrones consisten típicamente en secuencias del cromosoma bacteriano que codifican un ARN que no contiene información para formar proteínas (ARN no codificante), una retrotranscriptasa y una proteína efectora, generalmente tóxica. En condiciones normales, la retrotranscriptasa sintetiza una cadena de ADN utilizando parte del ARN no codificante como plantilla. La molécula mixta de ADN y ARN mantiene inactiva la toxina. El sistema se altera en caso de infección vírica imparable, la toxina se activa y la bacteria se suicida (Figura 2).
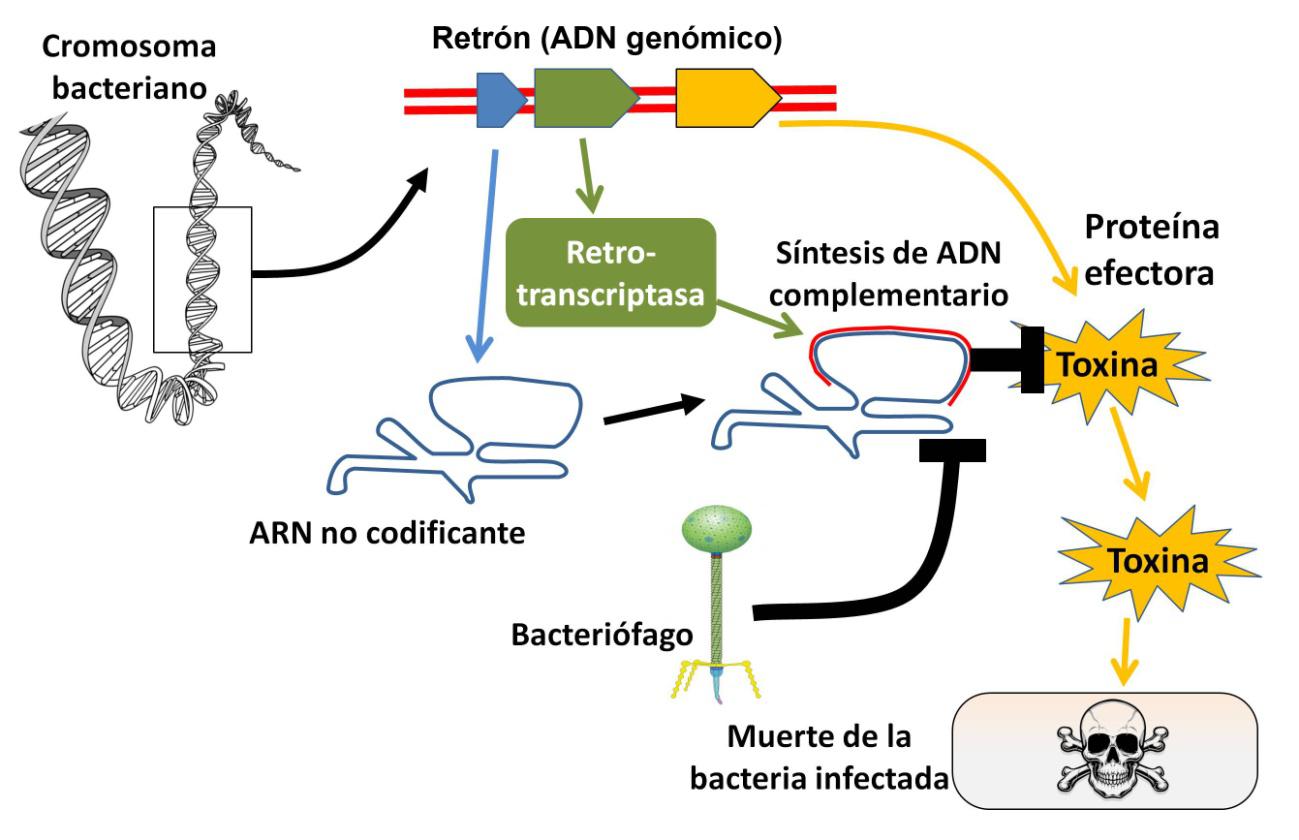
Se podría pensar, ¿qué clase de defensa antivírica implica un suicidio celular? La clave está en que la muerte de la bacteria impide la multiplicación del virus y su transmisión al resto de la población bacteriana. Esta estrategia “kamikaze” se conoce como infección abortiva y beneficia al conjunto de la población a expensas del individuo infectado.
Otras defensas basadas en retrotranscriptasas son las DRTs (defense-associated retrotranscriptases). Una de ellas, la DRT2, constituía hasta ahora un enigma ya que, a diferencia de los retrones, solo está formada por el ADN que genera la secuencia de ARN no codificante y una retrotranscriptasa, sin ninguna proteína efectora. Dos artículos publicados simultáneamente en Science el pasado mes de octubre desvelaron la función de DRT2, y han supuesto una auténtica conmoción en medios científicos. El sistema se basa en algo excepcional: la síntesis de un nuevo gen, ausente en el genoma bacteriano original.
Ambos grupos de investigación, en la Universidad Columbia de Nueva York y en el Instituto Tecnológico de Massachusetts, utilizaron el mismo modelo, la bacteria Klebsiella pneumoniae. Comprobaron que la expresión de DRT2 provocaba la síntesis del ARN no codificante, que se pliega sobre sí mismo, y la retrotranscriptasa (Figura 3). Como sucede con los retrones, esta enzima genera la cadena de ADN complementaria de una parte del ARN. Pero, y aquí viene la sorpresa, cuando termina la síntesis, se produce un “salto” y la enzima vuelve a empezar la síntesis del ADN desde el principio. Este ciclo se repite una y otra vez, generando una cadena sencilla de ADN con múltiples secuencias repetidas, a la que se denominó ADN concatenado.
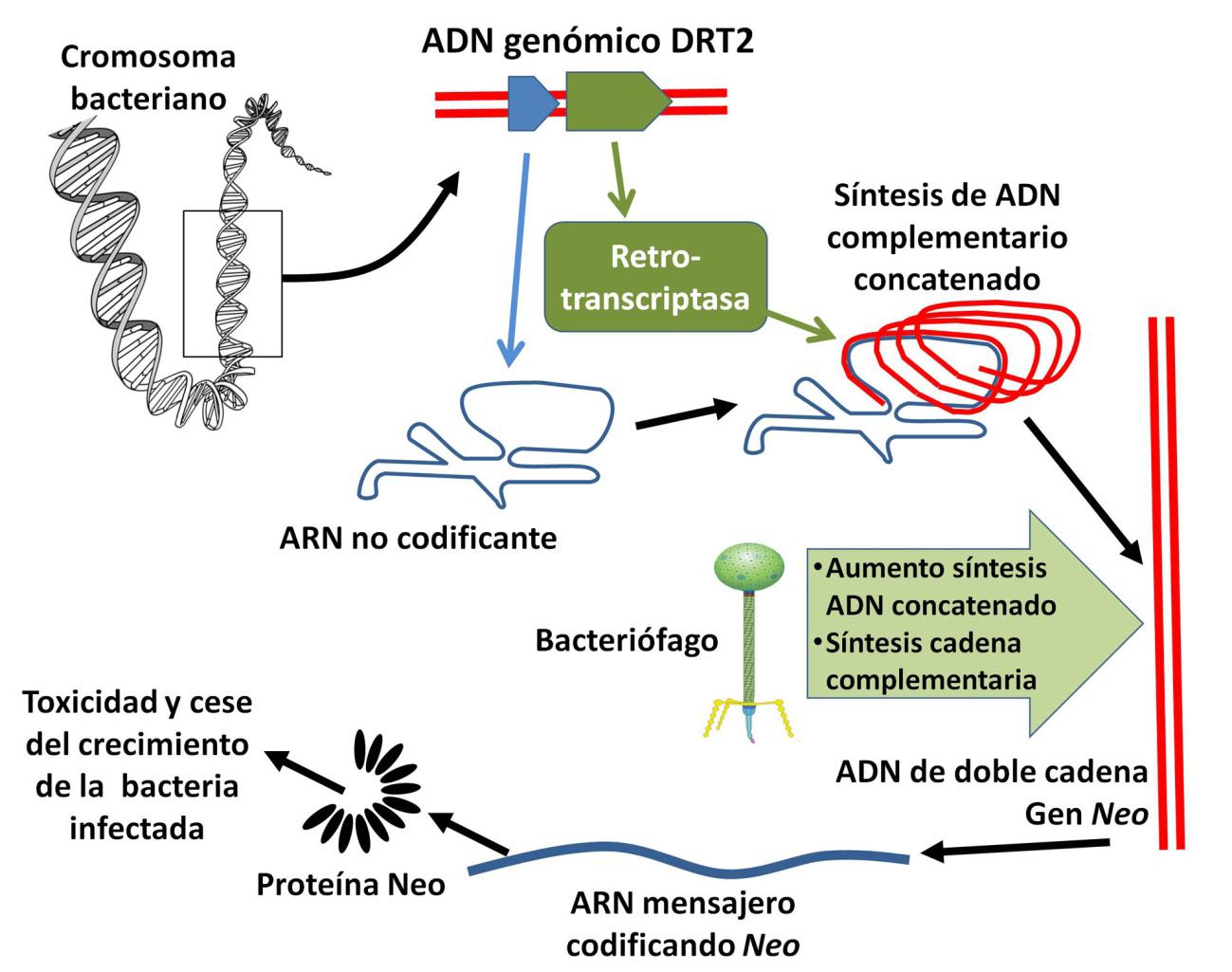
En caso de infección vírica suceden dos cosas (Figura 3). La síntesis de ADN concatenado aumenta miles de veces y, al mismo tiempo, se sintetiza la hebra complementaria, generando un ADN de doble cadena, como el del cromosoma bacteriano. Este ADN se comporta como un nuevo gen, denominado Neo1, que se traduce en una proteína con múltiples repeticiones de una secuencia de 40 aminoácidos. La predicción mediante Alphafold es que la proteína Neo está formada por un gran número de hélices. Esta proteína bloquea el crecimiento y la reproducción de la bacteria, evitando así la proliferación del virus.
Son muchas las incógnitas que permanecen, por ejemplo, qué tamaño puede alcanzar Neo, ya que la longitud de su gen no parece tener límites. Tampoco se sabe cómo Neo bloquea el crecimiento bacteriano. Lo que sí parece probable, según la comparación de secuencias que se ha hecho, es que este mecanismo esté presente en muchas otras bacterias.
Lo que resulta realmente llamativo de estos resultados es que a partir de un ARN no codificante se sintetice una secuencia de ADN que actúa como un gen extracromosómico que genera una proteína no codificada por el genoma bacteriano. Estamos, por tanto, ante un caso hasta ahora impensable de flujo de información genética entre el ADN y el ARN (Figura 1).
Referencias:
Tang, S., Conte, V., Zhang, D.J., et al. (2024). De novo gene synthesis by an antiviral reverse transcriptase. Science. Https://doi: 10.1126/science.adq0876.
Wilkinson, M.E., Li, D., Gao, A.F., et al. (2024). Phage-triggered reverse transcription assembles a toxic repetitive gene from a noncoding RNA. Science. Https://doi: 10.1126/science.adq3977.
Sobre el autor: Ramón Muñoz-Chápuli Oriol es Catedrático de Biología Animal (jubilado) de la Universidad de Málaga
Nota
1 Por never ending open reading frame, marco abierto de lectura sin fin. Un marco abierto de lectura es una secuencia de ADN con una señal de inicio de la traducción a proteína y una señal de terminación. El “sin fin” se debe a que, en este caso, la secuencia no tiene señales de terminación
El artículo Defensa insólita contra virus bacteriófagos: síntesis de un gen nuevo… y tóxico se ha escrito en Cuaderno de Cultura Científica.













































































